Kapitel 6 Datenaufbereitung
Datenaufbereitung (data wrangling) bezeichnet den Prozess, in dem Rohdaten so verändert, sortiert, umstrukturiert und ausgewählt werden, dass man sie für die anvisierte Analyse verwenden kann.
Im Einzelnen werden in diesem Kapitel die folgenden Funktionen erklärt:
filter()zur Auswahl von Fällenarrange()zur Sortierung von Fällenrec()zum Umcodieren von Variablenrow_means()undrow_sums()sowiemutate()zum Anlegen und Berechnen neuer Variablenselect()zur Auswahl von Variablensummarize()um Daten zu verdichten
Die letzte Funktion entfaltet besondere Stärken im Zusammenhang mit group_by(). Dadurch kann man Auswertungen oder bestimmte Datentransformationen nach Gruppen aufteilen.
Fast alle der hier vorgestellten Funktionen gehören zum Paket dplyr aus dem tidyverse. Die einzige Ausnahme bildet rec() aus dem Paket sjmisc. Obwohl sie aus unterschiedlichen Paketen stammen, folgen alle dem tidyverse-Konzept und funktionieren auf ähnliche Weise (vgl. Wickham and Grolemund 2017, Kap. 5.1.3):
Das erste Argument ist immer der Dataframe.
Die folgenden Argumente beschreiben, wie der Dataframe umgeformt werden soll (ohne Anführungsstriche).
Soll innerhalb der Funktionen auf Variablen aus dem Dataframe zugegriffen werden, kann man diese direkt ansprechen (also einfach nur
var_nameund nichtdata$var_nameoder"var_name").Das Ergebnis ist immer ein Dataframe.
6.1 Prerequisites
Als Datensatz dient in diesem Kapitel der “starwars”-Datensatz, der im Paket dplyr enthalten ist. Er enthält verschiedene Merkmale von Starwars-Figuren:
## # A tibble: 87 x 14
## name height mass hair_color skin_color eye_color birth_year sex gender
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
## 1 Luke S… 172 77 blond fair blue 19 male mascu…
## 2 C-3PO 167 75 <NA> gold yellow 112 none mascu…
## 3 R2-D2 96 32 <NA> white, bl… red 33 none mascu…
## 4 Darth … 202 136 none white yellow 41.9 male mascu…
## 5 Leia O… 150 49 brown light brown 19 fema… femin…
## 6 Owen L… 178 120 brown, grey light blue 52 male mascu…
## 7 Beru W… 165 75 brown light blue 47 fema… femin…
## 8 R5-D4 97 32 <NA> white, red red NA none mascu…
## 9 Biggs … 183 84 black light brown 24 male mascu…
## 10 Obi-Wa… 182 77 auburn, wh… fair blue-gray 57 male mascu…
## # … with 77 more rows, and 5 more variables: homeworld <chr>, species <chr>,
## # films <list>, vehicles <list>, starships <list>Außerdem werde ich auch auf den bereits bekannten Generation-Z-Datensatz zurückgreifen, weil dieser für das Umcodieren geeigneter ist.
In diesem Kapitel werden – wie oben beschreiben – die Pakete dplyr aus dem tidyverse sowie sjmisc genutzt. Jedes neue Paket, dass zum ersten Mal verwendet wird, muss natürlich wie im Abschnitt [#### Files, Plots, Packages, Help & Viewer] beschrieben installiert werden. Danach muss das Paket auch noch im Skript mit dem library-Befehl geladen werden. Dadurch weiß R, dass das Paket in der aktuellen Session verwendet werden soll und macht die Funktionen des Paketes verfügbar.
library(tidyverse)
library(sjmisc)6.2 Die Pipe
 Bevor es mit den einzelnen Schritten der Datenaufbereitung losgeht, wird an dieser Stelle noch ein neuer Operator eingeführt, die Pipe. In R geschrieben durch die Zeichenfolge
Bevor es mit den einzelnen Schritten der Datenaufbereitung losgeht, wird an dieser Stelle noch ein neuer Operator eingeführt, die Pipe. In R geschrieben durch die Zeichenfolge %>%. Eine Pipe kann man auch durch den Shortcut Ctrl/Strg + Shift + m einfügen. Merken Sie sich diesen Shortcut gut, Sie werden ihn oft brauchen!
Die Pipe macht etwas, das für Sie zunächst tendenziell unsinnig klingen muss: Sie leitet das Ergebnis einer Funktion als Argument an die nächste Funktion weiter. Gerade bei der Datenaufbereitung ist das jedoch sehr praktisch, weil man häufig mehrere Funktionen hintereinanderschalten muss: Man möchte z.B. zunächst ein paar Fälle herausfiltern, dann eine neue Variable bilden, alte Variablen löschen, andere Variablen umcodieren, dann Variablen auswählen, den Datensatz neu sortieren und schließlich nochmal ein paar Fälle herausfiltern und zum Schluss eine Analyse machen. Zusammengefasst: Es sollen sehr viele Transformationen eines Datensatzes hintereinander geschaltet werden.
6.2.1 Der Aufbau im Detail
Hier der schematische Aufbau einer Datentransformation mit der Pipe, damit Sie nachvollziehen können, wie der Pipe-Operator funktioniert (Achtung, jetzt folgt Pseudo-Code, der nur der Veranschaulichung dient und nicht 1:1 ausführbar ist):
new_data <- data %>%
transformation_1("do something") %>%
transformation_2("do something else") %>%
transformation_3("do something else else") Schauen wir uns mal zeilenweise an, was hier passiert:
Erste Zeile: Der Start
Zunächst wird ein neues Objekt
new_dataerzeugt, indem das alte Objektdata- also unser Datensatz - kopiert wird. Dieser Schritt ist immer dann nötig, wenn man mit dem Datensatz weiterarbeiten möchte.Nachdem die Operation durchgeführt wurde, wird das Ergebnis dieser Operation (also das neue Objekt
new_data) mit der Pipe%>%an die nächste Zeile übergeben.
Zweite Zeile: Wo landet das Objekt
new_data? Ich habe eben geschrieben, dass das Objekt an die nächste Zeile übergeben wurde. Es ist vielleicht etwas irritierend, dass es gar nicht mehr zu sehen ist. Also wo ist es?Es steckt in der Funktion dieser Zeile, also im
transformation_1()und zwar als erstes Argument. Durch die Pipe ist es quasi unsichtbar. Gedanklich kann man sich den Befehl in dieser Zeile so vorstellen:transformation_1(new_data, "do something")- nur, dass mannew_datadort nicht extra erwähnen muss, weil durch die Pipe in der vorhergehenden Zeile klar ist, dass dieses Objet das erste Argument ist.Die Funktion
transformation_1wird also mit den beiden Argumentennew_dataund"do something"ausgeführt. Der Datensatz verändert sich entsprechend. Er behält aber den gleichen Namen.Am Ende der Zeile steht wieder eine Pipe
%>%. Auch sie leitet das Ergebnis der vorhergehenden Transformation an die nächste Zeile weiter.
Dritte Zeile: …same procedure as every pipe…
Wieder landet der (nun einmal transformierte) Dataframe
new_dataals erstes Argument in einer Funktion, diesmal intransformation_2().Wieder wird der Dataframe irgendwie transformiert und heißt noch immer gleich.
Wieder wird er durch die Pipe am Ende der Zeile an die nächste Zeile übergeben.
Vierte Zeile: Das Ende naht.
Auch hier wieder dasselbe Spiel wie zuvor: Der Datensatz landet als erstes Argument in der Funktion
transformation_3(), die irgendwelche Operationen mit ihm durchführt.Nach der Transformation ist allerdings Schluss, denn da ist keine weitere Pipe. Der nun dreifach transformierte Datensatz ist jetzt fertig und liegt als neues Objekt
new_datavor. Sie finden es im Environment-Tab.
Insgesamt ist die Pipe-Schreibweise sehr übersichtlich, weil die einzelnen Transformationen schön untereinander aufgeführt werden. Man kann also sehr schnell erkennen, was mit dem Dataframe passiert.
Noch eine kleine Anmerkung zur ersten Zeile: Dort habe ich durch new_data <- data ein neues Objekt erzeugt. Das ist immer dann sinnvoll, wenn man nach der Transformation die Daten als Objekt vorliegen haben möchte, um damit z.B. verschiedene statistische Berechnungen durchzuführen. Manchmal benötigt man aber gar kein neues Objekt. Vielleicht möchte man nur temporär etwas ausgeben. In diesem Fall könnte man auch direkt mit data %>% starten. In diesem Kapitel werde ich beides benutzen, da es mir hier auch nicht immer darum geht, den Datensatz tatsächlich zu transformieren.
6.2.2 Schlechtere Alternativen zur Pipe
Schauen wir uns einmal an, was die Alternativen zur Arbeit mit der Pipe wären. Es gibt 3:
Selbstverständlich könnte man alle Datentransformationen nacheinander machen und dabei den Dataframe, den es zu bearbeiten gilt, immer wieder überschreiben. Das ist jedoch keine saubere Arbeitsweise, es ist sehr anfällig für Fehler.
Eine andere Option wäre es, jedes Mal ein neues Objekt zu erzeugen und die Objekte dann durchzunummerieren oder zu benennen (
data_1,data_2,data_3oderdata_filtered,data_sorted,data_with_var_x). Auch nicht sehr übersichtlich und ebenfalls fehleranfällig.Die dritte Möglichkeit wäre es, Funktionen ineinander zu verschachteln, etwa so:
fun1(fun2(fun3(arg1, arg2)), arg1, arg2). R würde diese dann von innen nach außen abarbeiten. Das ist zwar sehr kompakt, allerdings ist es sehr schwer, hier den Überblick zu behalten und auch hier sind Fehler (etwa bei der Klammersetzung) vorprogrammiert.
Besser sie gewöhnen sich die Arbeit mit der Pipe direkt an. Gerade für den Bereich Datenaufbereitung macht die Pipe sehr viel Sinn, weil in den Funktionen das Datenargument immer an der ersten Stelle steht. Das kommt der Pipe sehr entgegen, weil man den Dataframe so quasi von oben nach unten durch die Pipe leiten und in jedem Schritt ein bisschen weiter umformen kann. Auch wenn die Pipes in diesem Kapitel noch nicht besonders lang sein werden, verwende ich diese Schreibweise – einfach, damit Sie sich daran gewöhnen.
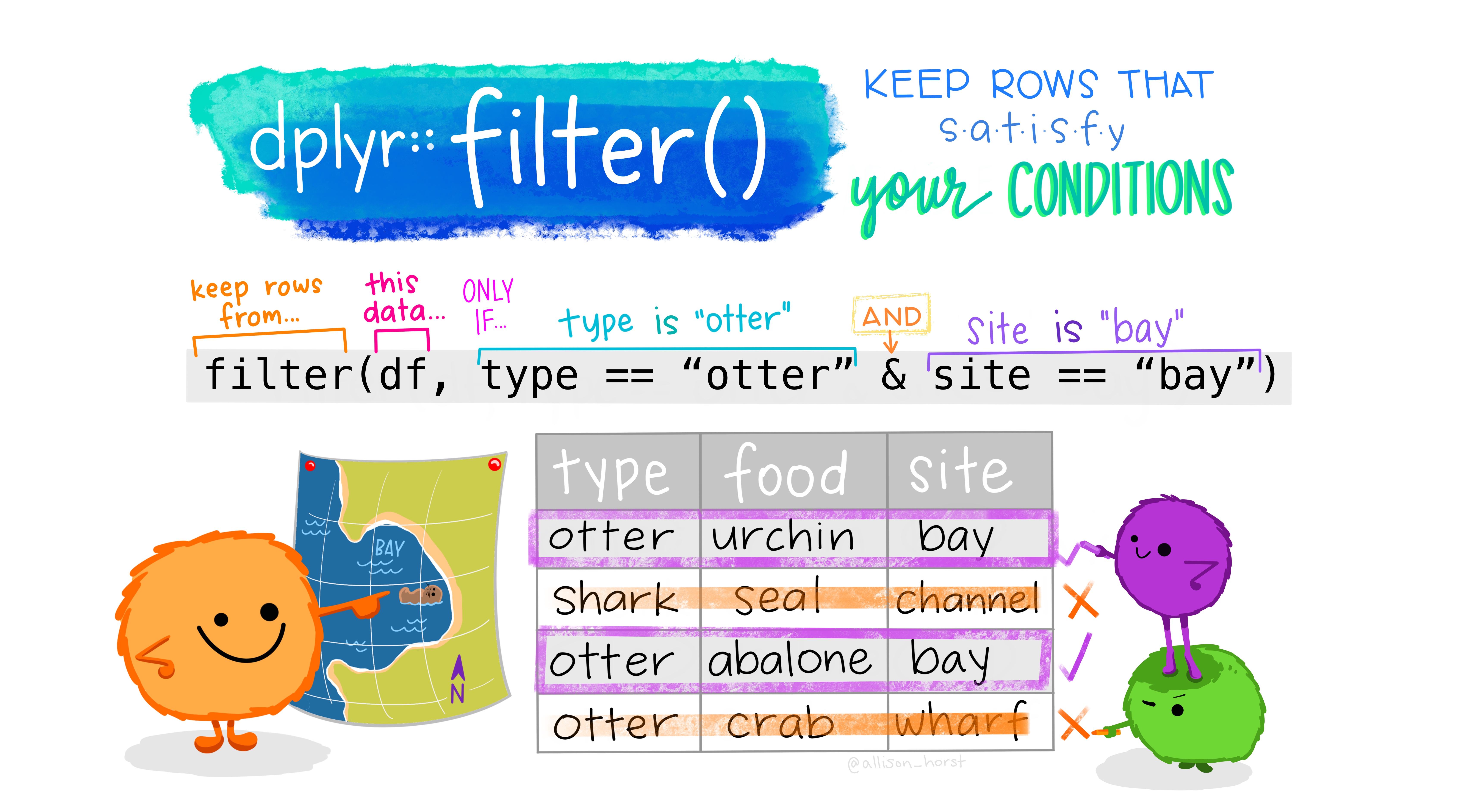
6.3 Filter: Fälle auswählen
Mit Filtern kann man die Fallzahl eines Datensatzes nach bestimmten Kriterien verringern, also Fälle herausfiltern, die man nicht benötigt bzw. momentan nicht berücksichtigen möchte.
Fälle entfernen, die man grundsätzlich nicht im Datensatz haben wollte, z.B. Minderjährige, wenn man nur Erwachsene befragen wollte.
Dubletten entfernen (falls aus Versehen ein Fall doppelt eingegeben wurde)
Einen Datensatz für eine bestimmte Analyse erstellen, die sich nur auf eine Teilstichprobe bezieht:
alle Folgen von Serien die länger als 60 Minuten sind
nur nicht-männliche Befragte
alle Personen die YouTube oder Instagram regelmäßig nutzen
Im folgenden Beispiel möchte ich einen Starwars-Datensatz erstellen, der nur Fälle von Figuren enthält, deren Körpergröße mindestens bei 200 cm liegt. Bevor man einen Filter anwendet, sollte man sich aber zunächst einen Überblick über die Ausgangslage verschaffen. Ich lasse mir deshalb einmal die Anzahl der Zeilen im Datensatz ausgeben und schaue mir die ersten paar Fälle an:
nrow(starwars)## [1] 87head(starwars)## # A tibble: 6 x 14
## name height mass hair_color skin_color eye_color birth_year sex gender
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
## 1 Luke Sk… 172 77 blond fair blue 19 male mascu…
## 2 C-3PO 167 75 <NA> gold yellow 112 none mascu…
## 3 R2-D2 96 32 <NA> white, bl… red 33 none mascu…
## 4 Darth V… 202 136 none white yellow 41.9 male mascu…
## 5 Leia Or… 150 49 brown light brown 19 fema… femin…
## 6 Owen La… 178 120 brown, grey light blue 52 male mascu…
## # … with 5 more variables: homeworld <chr>, species <chr>, films <list>,
## # vehicles <list>, starships <list>Okay, der ursprüngliche Datensatz hat 87 Zeilen (Starwars-Charactere) und bei der Körpergröße “height” gibt es gemischte Werte (über und unter 200 cm).
Als nächstes muss eine Filterbedingung festgelegt werden. Die Filterbedingung ist nach den Daten das zweite und zwingende Argument, dass die filter()-Funktion benötigt. Hier kommt der Datentyp “logical” ins Spiel, den wir hier besprochen haben. Anhand der Filterbedingung prüft die Funktion filter() für jeden Fall im Datensatz, ob eine zuvor von uns definierte Bedingung TRUE oder FALSE ist. Ist das Ergebnis der Prüfung TRUE verbleibt der Fall im Datensatz. Ist es FALSE wird der Fall aus dem Datensatz entfernt. Die Prüfung erfolgt anhand der relationalen Operatoren (z.B. == für “ist gleich,” != für “ist ungleich” oder < für “ist kleiner als”).
Im Beispiel wollen wir Starwars-Figuren die eine Mindestgröße von 200 überschreiten in einem Datensatz abspeichern. Wir müssen also die Bedingung “Die Größe ist mindestens 200 cm” so formulieren, dass R sie versteht. Das geht mit der Bedingung height >= 200:
data_tall <- starwars %>%
filter(height >= 200)Gar nicht so schwer, aber hat das auch funktioniert? Schauen wir uns nochmal die Fallzahl und den Datensatz genauer an:
nrow(data_tall)## [1] 11head(data_tall)## # A tibble: 6 x 14
## name height mass hair_color skin_color eye_color birth_year sex gender
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
## 1 Darth V… 202 136 none white yellow 41.9 male mascul…
## 2 Chewbac… 228 112 brown unknown blue 200 male mascul…
## 3 IG-88 200 140 none metal red 15 none mascul…
## 4 Roos Ta… 224 82 none grey orange NA male mascul…
## 5 Rugor N… 206 NA none green orange NA male mascul…
## 6 Yarael … 264 NA none white yellow NA male mascul…
## # … with 5 more variables: homeworld <chr>, species <chr>, films <list>,
## # vehicles <list>, starships <list>Tatsächlich! Im Datensatz sind jetzt nur noch n = 11 Fälle und in der Variable height haben alle den Wert 200 oder einen höheren Wert.
Natürlich kann man in R auch auf nominale Variablen filtern, z.B. auf eine bestimmte Augenfarbe. Im folgenden Datensatz speichere ich alle Starwars-Figuren ab, die orangene Augen haben. Dafür benötige ich die Filterbedingung: eye_color == "orange". Man braucht hier zwingend doppelte Gleichzeichen. Dies ist nötig, weil das einfache Gleichzeichen von R als Zuweisungsoperator <- verstanden würde. Hier soll aber nichts zugewiesen, sondern lediglich etwas verglichen werden. Beachten Sie außerdem die Anführungszeichen. Wir brauchen Sie, weil es sich um eine Text-Variable (character) handelt.
data_orange <- starwars %>%
filter(eye_color == "orange")Und Kontrolle:
nrow(data_orange)## [1] 8head(data_orange)## # A tibble: 6 x 14
## name height mass hair_color skin_color eye_color birth_year sex gender
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
## 1 Jabba … 175 1358 <NA> green-tan,… orange 600 herma… mascu…
## 2 Ackbar 180 83 none brown mott… orange 41 male mascu…
## 3 Jar Ja… 196 66 none orange orange 52 male mascu…
## 4 Roos T… 224 82 none grey orange NA male mascu…
## 5 Rugor … 206 NA none green orange NA male mascu…
## 6 Sebulba 112 40 none grey, red orange NA male mascu…
## # … with 5 more variables: homeworld <chr>, species <chr>, films <list>,
## # vehicles <list>, starships <list>Perfekt! Jetzt machen wir es komplizierter. Wir möchten jetzt alle Personen haben, die orange oder gelbe Augen haben und größer als 200 cm sind. Um eine so komplexe Bedingung zu formulieren, braucht man neben den relationalen Operatoren auch noch logische Operatoren und Klammer-Regeln.
Mit logischen Operatoren kann man Bedingungen verknüpfen oder gegenseitig ausschließen. Die Wichtigsten sind:
&für “und”|für “oder”!für “nicht”
Die Bedingung “orange oder gelbe Augen und von Tatooine” lässt sich also wie folgt formulieren: (eye_color == "orange" | eye_color == "yellow") & height > 200. Hier kommt es haargenau auf die Klammern an. Wären sie nicht gesetzt würde R möglicherweise orange-äugigen (egal welche Körpergröße) und alle gelb-äugigen mit Körpergröße über 200 cm in den Dataframe packen.
data_filter <- starwars %>%
filter((eye_color == "orange" | eye_color == "yellow") & height > 200)
nrow(data_filter)## [1] 4head(data_filter)## # A tibble: 4 x 14
## name height mass hair_color skin_color eye_color birth_year sex gender
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
## 1 Darth V… 202 136 none white yellow 41.9 male mascul…
## 2 Roos Ta… 224 82 none grey orange NA male mascul…
## 3 Rugor N… 206 NA none green orange NA male mascul…
## 4 Yarael … 264 NA none white yellow NA male mascul…
## # … with 5 more variables: homeworld <chr>, species <chr>, films <list>,
## # vehicles <list>, starships <list>Ein häufiger Use-Case für Filter, der bisher noch nicht angesprochen wurde, ist es, fehlende Werte aus den Daten herauszufiltern. Das folgende Codebeispiel sortiert Fälle aus, die in der Variable height einen fehlenden Wert (NA) haben:
data_filter_na <- starwars %>%
filter(!is.na(height))
nrow(data_filter_na)## [1] 81head(data_filter_na)## # A tibble: 6 x 14
## name height mass hair_color skin_color eye_color birth_year sex gender
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
## 1 Luke Sk… 172 77 blond fair blue 19 male mascu…
## 2 C-3PO 167 75 <NA> gold yellow 112 none mascu…
## 3 R2-D2 96 32 <NA> white, bl… red 33 none mascu…
## 4 Darth V… 202 136 none white yellow 41.9 male mascu…
## 5 Leia Or… 150 49 brown light brown 19 fema… femin…
## 6 Owen La… 178 120 brown, grey light blue 52 male mascu…
## # … with 5 more variables: homeworld <chr>, species <chr>, films <list>,
## # vehicles <list>, starships <list>6.4 Arrange: Fälle sortieren
Mit arrange() lassen sich Fälle in einem Datensatz sortieren. Die Sortierung sollte zwar auf statistische Analysen keinen Einfluss haben, aber dennoch ist dieses Feature nützlich, wenn man z.B. Tabellen hübsch formatieren möchte.
Der Einsatz von arrange() ist sehr simpel. Man muss der Funktion nach dem Datensatz lediglich die Variable übergeben, nach der sortiert werden soll, hier z.B. nach der Körpergröße:
# aufsteigend sortieren
starwars %>%
arrange(height) %>%
head()## # A tibble: 6 x 14
## name height mass hair_color skin_color eye_color birth_year sex gender
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
## 1 Yoda 66 17 white green brown 896 male mascu…
## 2 Ratts Ty… 79 15 none grey, blue unknown NA male mascu…
## 3 Wicket S… 88 20 brown brown brown 8 male mascu…
## 4 Dud Bolt 94 45 none blue, grey yellow NA male mascu…
## 5 R2-D2 96 32 <NA> white, bl… red 33 none mascu…
## 6 R4-P17 96 NA none silver, r… red, blue NA none femin…
## # … with 5 more variables: homeworld <chr>, species <chr>, films <list>,
## # vehicles <list>, starships <list>Die Daten sind jetzt aufsteigend sortiert. Um eine absteigende Sortierung zu erreichen, benötigen wir die Hilfe von desc(). Das sieht dann so aus:
# absteigend sortieren
starwars %>%
arrange(desc(height))%>%
head()## # A tibble: 6 x 14
## name height mass hair_color skin_color eye_color birth_year sex gender
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
## 1 Yarael… 264 NA none white yellow NA male mascu…
## 2 Tarfful 234 136 brown brown blue NA male mascu…
## 3 Lama Su 229 88 none grey black NA male mascu…
## 4 Chewba… 228 112 brown unknown blue 200 male mascu…
## 5 Roos T… 224 82 none grey orange NA male mascu…
## 6 Grievo… 216 159 none brown, whi… green, ye… NA male mascu…
## # … with 5 more variables: homeworld <chr>, species <chr>, films <list>,
## # vehicles <list>, starships <list>Selbstverständlich kann man auch nach mehreren Variablen sortieren und dabei aufsteigende und absteigende Sortierung nach Belieben mischen:
# nach mehreren Variablen sortieren
starwars %>%
arrange(sex, hair_color, desc(height))%>%
head()## # A tibble: 6 x 14
## name height mass hair_color skin_color eye_color birth_year sex gender
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
## 1 Mon Mot… 150 NA auburn fair blue 48 fema… femin…
## 2 Luminar… 170 56.2 black yellow blue 58 fema… femin…
## 3 Barriss… 166 50 black yellow blue 40 fema… femin…
## 4 Shmi Sk… 163 NA black fair brown 72 fema… femin…
## 5 Zam Wes… 168 55 blonde fair, gree… yellow NA fema… femin…
## 6 Beru Wh… 165 75 brown light blue 47 fema… femin…
## # … with 5 more variables: homeworld <chr>, species <chr>, films <list>,
## # vehicles <list>, starships <list>6.5 Select: Variablen auswählen
Die Funktion select() dient genau wie filter() dazu, den Datensatz zu verkleinern. Jedoch geht es bei select() darum, Variablen auszuwählen. Dazu muss man die Variablen, die im Datensatz verbleiben sollen, einfach an die Funktion übergeben. Alle anderen Variablen, die nicht vorkommen, werden gelöscht.
# Variablen auswählen
starwars %>%
select(name, homeworld, species) %>%
head()## # A tibble: 6 x 3
## name homeworld species
## <chr> <chr> <chr>
## 1 Luke Skywalker Tatooine Human
## 2 C-3PO Tatooine Droid
## 3 R2-D2 Naboo Droid
## 4 Darth Vader Tatooine Human
## 5 Leia Organa Alderaan Human
## 6 Owen Lars Tatooine HumanWill man nur einzelne Variablen löschen, so geht dies mit einem - vor dem Variablennamen. select(data, -birth_year) löscht also das Alter, alle anderen Variablen würden aber erhalten bleiben.
Es gibt auch die Möglichkeit, Variablen auszuwählen, die einem bestimmten Schema entsprechen, z.B. deren Name mit “var_name_” beginnt. Die Syntax dafür ist starts_with("var_name_"). Ähnlich kann man auch Variablen in einem bestimmten Bereich auswählen, also alle von var_name_1 bis var_name_x. Dafür müsste man beispielsweise height:eye_color eingeben.
Zudem kann man select() auch dazu verwenden, die Variablen im Datensatz umzusortieren. Dazu schreibt man die Variablen einfach in der neuen Reihenfolge in die Funktion. Beim Umsortieren gibt es ebenfalls einige nützliche Helfer. Einer ist beispielsweise die Funktion everything() - quasi ein Alias für alle Variablen die bis dahin noch nicht genannt wurden.
# Variablen neu sortieren
starwars %>%
select(name, homeworld, everything()) %>%
head()## # A tibble: 6 x 14
## name homeworld height mass hair_color skin_color eye_color birth_year sex
## <chr> <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr>
## 1 Luke … Tatooine 172 77 blond fair blue 19 male
## 2 C-3PO Tatooine 167 75 <NA> gold yellow 112 none
## 3 R2-D2 Naboo 96 32 <NA> white, bl… red 33 none
## 4 Darth… Tatooine 202 136 none white yellow 41.9 male
## 5 Leia … Alderaan 150 49 brown light brown 19 fema…
## 6 Owen … Tatooine 178 120 brown, gr… light blue 52 male
## # … with 5 more variables: gender <chr>, species <chr>, films <list>,
## # vehicles <list>, starships <list>6.6 Variablen umcodieren
Eine häufige Aufgabe bei der Datenaufbereitung ist das Umcodieren. Beim Umcodieren wird das Wertespektrum einer Variable verändert oder verdichtet. Ein Anwendungsfall wäre es, stetige Variablen damit in Kategorien einteilen (z.B. Altersgruppen bilden). Ein weiterer Anwendungsfall sind Variablen, die “falsch herum” codiert wurden und jetzt gedreht werden müssen. In dem Generation-Z-Datensatz sind beispielsweise die Variablen zu “Verbundenheit” unintuitiv codiert: Ein niedriger Zahlenwert entspricht einer hohen Verbundenheit. Der Wert 1 hat das Werte-Label “sehr verbunden,” der Wert 5 ist hingegen mit “überhaupt nicht verbunden” codiert. Sie können das im Codebuch sehen, aber das folgende Skript verdeutlicht diesen Umstand an der Variable verbundenheit_europa.
library(sjlabelled)
# einen Vektor mit den Werten einer Variable erzeugen
values = get_values(data$verbundenheit_europa)
# einen Vektor mit den Labels einer Variable erzeugen
labels = get_labels(data$verbundenheit_europa)
cbind(values, labels) # beide Vektoren zusammenbinden## values labels
## [1,] "1" "Sehr verbunden"
## [2,] "2" "Ziemlich verbunden"
## [3,] "3" "Nicht sehr verbunden"
## [4,] "4" "Überhaupt nicht verbunden"
## [5,] "99" "Weiß nicht"Intutiver wäre es, wenn mit einem hohen Zahlenwert auch eine große Verbundeheit einher ginge. Bei den gelabelten Daten, die hier vorliegen, geht das Umcodieren sehr gut über den Befehl rec() aus dem Paket sjmisc. Ein Tipp für SPSS-Umsteiger: Der Befehl ist sehr stark an die Logik von SPSS angelehnt.
Der rec()-Befehl fügt sich in die tidyverse-Logik ein und erwartet als erstes Argument genau wie die dplyr-Funktionen den Dataframe. Deshalb kann man den Befehl ebenfalls sehr gut in der Pipe einsetzen. Das zweite Argument ist die Variable, die umcodiert werden soll. Man kann hier auch mehrere Variablen einsetzen, in unserem Fall alle die mit verbundenheit_ beginnen. Ein kleiner Einschub: An dieser Stelle wird bereits deutlich, dass Variablennamen möglichst so zu vergeben sind, dass Variablen eines Konzeptes immer gleich benannt werden. Eine reine Nummerierung von Variablen würde den Befehl erheblich länger machen.
Das letzte und entscheidende Argument ist die Anweisung zur Umcodierung. Es heißt rec und beinhaltet einen Text mit den Anweisungen in der Form "werte_label = neuer_wert". Getrennt durch ein Semikolon kann man auch mehrere Anweisungen gleichzeitig übergeben. Jede geplante Umcodierung muss explizit genannt werden. Sollte ein oder mehrere Werte nicht von der Umcodierung betroffen sein, kann man die “restlichen” Werte durch ein "else=copy" auffangen. Dadurch wird der Wert aus der ursprünglichen Variable einfach in die neue kopiert. In unserem Beispiel betrifft das den Wert 99 = “weiß nicht.” Die 99 soll ganz unabhängig von der Umcodierung immer diesen Wert beibehalten.
Die Funktion rec() erzeugt neue Variablen, die den gleichen Namen haben wie die ursprünglichen, ergänzt um ein _r am Ende. Diese Endung soll deutlich machen, dass es sich um die recodierte Variante der Variablen handelt.
library(sjmisc)
data <- data %>%
rec(starts_with("verbundenheit_"), rec = "Sehr verbunden = 4;
Ziemlich verbunden = 3;
Nicht sehr verbunden = 2;
Überhaupt nicht verbunden = 1;
else=copy")
# Beispielhaft die Variable verbundenheit_dtl inklusive recodierter Variante anzeigen:
data %>%
select(starts_with("verbundenheit_dtl")) %>%
head()## verbundenheit_dtl verbundenheit_dtl_r
## 1 2 3
## 2 2 3
## 3 2 3
## 4 2 3
## 5 3 2
## 6 4 1Es ist immer ratsam, im Anschluss zu kontrollieren, ob die Umcodierung auch wie erwartet funktioniert hat. Dies kann z.B. über eine Kreuztabelle geschehen (vgl. Kapitel Kreuztabellen) oder wie hier durch ein “nebeneinanderlegen” der beiden Variablen.
Eine kleine Ergänzung noch. Ich habe den Datensatz hier über das sjlabelled-Paket in R hinein geladen: Selbstverständlich funktioniert rec() auch mit nicht-gelabelten Daten oder Daten, die durch das haven-Paket eingelesen wurden. In diesem Fall wären einfach die ursprünglichen Werte statt der (nicht vorhandenen) Wertelabels einzutragen: "1=4;2=3;3=2;4=1;else=copy"
Hier noch ein Beispiel mit dem Starwars-Datensatz, in dem die Variable für die Körpergröße in drei Gruppen eingeteilt wird:
sw_age_grp <- starwars %>%
rec(height, rec = "1:150 = small;
151:190 = medium;
190:900 = tall;
else=NA")6.7 Variablen berechnen
Es gibt viele unterschiedliche Wege, wie man in R neue Variablen berechnen kann. Wenn man Berechnungen nur unter bestimmten Bedingungen durchführen möchte, dann kann das Ganze auch ziemlich schnell sehr komplex werden.
Für den Einstieg habe ich hier zwei Wege herausgesucht. Einmal zur Bildung von Indices das sjmisc-Paket und aus dem tidyverse die Funktion mutate().
6.7.1 Summen und Mittelwertindices
Indices zu berechnen ist eine häufige Task bei der Datenaufbereitung. Zwei besonders häufige Formen sind:
Der Summenindex, bei dem die Werte mehrerer Variablen einfach aufsummiert werden (z.B. Anzahl genutzer Webseiten, Gesamtmediennutzungsdauer in Minuten)
Der Mittelwertindex, bei dem ein Mittelwert über mehrere Variablen hinweg gebildet wird.
Für diese beiden Index-Arten hält das sjmisc-Paket zwei interessante Funktionen bereit row_sums() und row_means().
Ich demonstriere im Folgenden die row_means()-Funktion, aber row_sums() funktioniert vom Prinzip her gleich. Ich bleibe dazu beim Generation-Z-Datensatz. Ich möchte jetzt für die 5 Verbundenheits-Variablen einen Mittelwertindex berechnen (ob das inhaltlich super sinnvoll ist, sei mal dahingestellt…).
Der Einsatz der Funktion sieht wie folgt aus:
gen_z_df_mean <- data %>%
row_means(verbundenheit_stadt_r:verbundenheit_europa_r, n = 4, var = "verbundenheit_r_mx")
head(gen_z_df_mean$verbundenheit_r_mx)## [1] 3,0 NA 2,6 3,0 2,2 1,6Neben dem Datensatz-Argument, welches hier wie gehabt über die Pipe übergeben wird, benötigt die Funktion row_means() noch weitere Argumente:
Die Variablen, die in dem Index zusammengefasst werden sollen
Das Argument
n =, in diesem Argument wird festgelegt, in wie vielen der Ursprungs-Variablen ein Fall einen gültigen Wert aufweisen muss, damit ein Index berechnet werden kann. Ich habe den Wert hier auf 4 gesetzt. Ein Befragter muss also mindestens 4 der 5 Variablen ausgefüllt haben, damit der Mittelwertindex berechnet wird.Optional das Argument
var =, das den Namen für den neuen Index in Anführungsstrichen enthält. Übergibt man dieses Argument nicht, wird der Index von R “rowmeans” genannt.
In der letzten Zeile lasse ich mir die ersten paar der errechneten Werte für die neue Variable/den neuen Mittelwertindex anzeigen.
6.7.2 Berechnen mit dplyr::mutate()
Mit mutate() kann man neue Variablen bilden und zwar nach beliebigen Formeln. Die Syntax dazu folgt dem Schema new_var_name = some calculation.
Im nächsten Code-Beispiel wird der Bodymass-Index der Starwars-Figuren berechnet.
Die Formel für den BMI ist: Gewicht durch Größe in Metern zum Quadrat.
Da die Größe dafür in Metern angegeben sein muss, im Starwars-Datensatz aber nur cm erfasst sind, müssen wir zusätzlich auch noch die Zentimeter in Meter umrechnen.
Damit wir die Daten im Anschluss an die Berechnung schön vergleichen können, wähle ich die beteiligten Variablen nach der Berechnung aus und sortiere nach dem BMI.
# BMI berechnen
starwars %>%
mutate(bmi = mass / (height/100)^2) %>%
select(name:mass, bmi) %>%
arrange(desc(bmi))## # A tibble: 87 x 4
## name height mass bmi
## <chr> <int> <dbl> <dbl>
## 1 Jabba Desilijic Tiure 175 1358 443.
## 2 Dud Bolt 94 45 50.9
## 3 Yoda 66 17 39.0
## 4 Owen Lars 178 120 37.9
## 5 IG-88 200 140 35
## 6 R2-D2 96 32 34.7
## 7 Grievous 216 159 34.1
## 8 R5-D4 97 32 34.0
## 9 Jek Tono Porkins 180 110 34.0
## 10 Darth Vader 202 136 33.3
## # … with 77 more rowsJetzt kennen Sie den BMI von Jabba the Hutt! Aber auch der BMI von Yoda ist ganz schön bedenklich…
6.7.3 Variablen unter einer Bedingung berechnen
Man kann natürlich auch Variablen anhand von logischen Ausdrücken berechnen, also eine Art Filterbedingung dafür zu Rate ziehen, welchen Wert die Variable annehmen soll. Es muss dafür wieder mit logischen Ausdrücken gearbeitet werden und wir brauchen eine Funktion die ìfelse() heißt. Die Funktion bekommt drei Argumente:
Den logischen Ausdruck bei dem für jeden Fall zu prüfen ist, ob er für diesen Fall
TRUEoderFALSEist.Einen Wert, den die Variable annehmen soll, wenn der Fall
TRUEeintritt.Einen Wert, den die Variable annehmen soll, wenn der Fall
FALSEeintritt.
Als Beispiel möchte ich eine Variable berechnen die 1 ist, wenn die Verbundenheit zu Europa größer ist, als die zu Deutschland und ansonsten 0. Ich nenne sie sieht_sich_als_europaeer.
# Variable berechnen mit Bedingung
data_eu <- data %>%
mutate(sieht_sich_als_europaeer = ifelse(verbundenheit_europa > verbundenheit_dtl, 1, 0))
# Für die Kontrolle relevante Variablen auswählen
data_eu %>%
select(lfdn, verbundenheit_europa, verbundenheit_dtl, sieht_sich_als_europaeer) %>% head()## lfdn verbundenheit_europa verbundenheit_dtl sieht_sich_als_europaeer
## 1 1634 2 2 0
## 2 1636 3 2 1
## 3 1637 2 2 0
## 4 1638 3 2 1
## 5 1639 3 3 0
## 6 1640 4 4 0
6.8 Summarize: Daten verdichten
Die letzte dplyr-Funktion, auf die ich hier eingehen möchte, ist summarize(). Im ersten Moment wirkt summarize() vielleicht ein bisschen wie eine komplizierte Art, deskriptiven Statistiken zu berechnen. Die Funktion kann aber viel mehr und das Entscheidende ist, dass sie nicht wie die im Kapitel “Deskriptive Statistiken” vorgestellten Funktionen einfach nur einen Kennwert zurückgibt, sondern einen Datensatz mit dem Ergebnis.
Möglicherweise werden Sie die Funktion zunächst kaum benutzen, aber später wiederentdecken. Der Vollständigkeit halber wird sie trotzdem an dieser Stelle kurz erläutert.
Im ersten Beispiel möchte ich den Mittelwert für Körpergröße der Starwars-Figuren ausrechnen, das haben wir ja schon mal gemacht. Aber jetzt eben mit der summarize()-Funktion.
# Test der summarize-Funktion
starwars %>%
summarise(mean_height = mean(height, na.rm = TRUE))## # A tibble: 1 x 1
## mean_height
## <dbl>
## 1 174.Das Ergebnis ist ein Datensatz, der eine neue Variable enthält, die mean_height heißt und nur einen Fall hat. Soweit so unspannend.
Das Geschickte an summarize() ist, dass die Funktion perfekt mit group_by() zusammenarbeitet. Mit group_by() kann man einen Dataframe aufteilen, so dass er dann wie mehrere getrennte Datensätze behandelt wird. Wir könnten also Gruppen bilden und die Anteile in diesen Gruppen rein deskriptiv vergleichen. Mich interessiert beispielsweise, ob es regionale Unterschiede bei der Größe der Charaktere gibt. Vergleichen wir mal Tatooine und Naboo. Zusätzlich lasse ich noch die Fallzahl der Gruppen mit ausgeben (n = n()):
# summarize mit filter & group_by
starwars %>%
filter(homeworld == "Tatooine" | homeworld == "Naboo") %>%
group_by(homeworld) %>%
summarize(mean = mean(height, na.rm = TRUE), n = n())## # A tibble: 2 x 3
## homeworld mean n
## <chr> <dbl> <int>
## 1 Naboo 175. 11
## 2 Tatooine 170. 10Natürlich funktioniert das nicht nur mit dem arithmetischen Mittel. Auch andere Berechnungen wären hier denkbar. Einige nützliche Funktionen finden Sie in der Hilfe von summarize().
Wichtige Funktionen aus diesem Kapitel
| Funktion | Paket | Beschreibung | Bemerkung |
|---|---|---|---|
%>% |
tidyverse/magrittr | Pipe-Operator | |
filter |
tidyverse/dplyr | Fälle auswählen | Filterbedingung mitrelationalen und logischen Operatoren |
arrange() |
tidyverse/dplyr | Sortieren | |
arrange(desc()) |
tidyverse/dplyr | Absteigend sortieren | |
select() |
tidyverse/dplyr | Variablen auswählen oder umsortieren | Selection Helpers |
rec() |
sjmisc | Variablen recodieren | Recodieranweisung als Text |
row_sums() |
sjmisc | Summenindex berechnen | n, var |
row_means() |
sjmisc | Mittelwertindex berechnen | n, var |
mutate() |
tidyverse/dplyr | Variablen berechnen | |
summarize()) |
tidyverse/dplyr | Daten aggregieren | |
group_by() |
tidyverse/dplyr | Daten aufteilen |